2023年12月9日,《Nature Communications》发表题为“Single cell multi-omics reveal intra-cell-lineheterogeneity across human cancer cell lines”的研究成果:科研团队利用DNBelab C4结合染色质可及性测序(scATAC-seq)分析了40个癌症细胞系,深入探索了各个癌症细胞系内的转录组和表观遗传异质性,发现转录组异质性是不同癌症的共同特征,并揭示了转录组和表观基因组之间相互作用的分子机制。此外,还发现了低氧环境可以重塑癌症细胞系的转录组异质性。该研究为了解癌症细胞系内异质性的潜在机制及其在人类癌症细胞系中的动态变化提供了有价值的见解,为未来以细胞系为基础的癌症研究提供了宝贵的单细胞多组学资源。

同时,研究团队整合相关数据联合深圳国家基因库共同搭建在线分析网站:人类癌症细胞系数据库(SCAtlas HCL),于国家基因库生命大数据平台(CNGBdb)同步上线,开放相关资源给全球科研同行使用。此外,该研究的相关数据均存储于国家基因库生命大数据平台(CNGBdb),项目编号为:CNP0004330 和 CNP0003658。
识别下方二维码可访问SCAtlas HCL。

db.cngb.org/cdcp/scatlashcl/
研究背景
过去的研究已经证实癌细胞之间存在着巨大的异质性,这是导致癌症进展、治疗耐药性和癌症复发的重要原因之一。然而,大多数研究都集中在不同癌症患者之间的异质性,而对于同一癌症患者内部的单细胞层面的异质性了解较少。因此,该研究旨在通过单细胞多组学技术来揭示人类癌症细胞系内的异质性,并可能为癌症治疗提供新的策略和目标。
研究样本
利用单细胞建库平台DNBelab C4结合scATAC-seq技术,分析了40个人类癌症细胞系样本(分布在9个谱系中,以实体瘤为主),以及2个人类正常细胞系样本(对照),涵盖了包括头颈癌、结直肠癌、肺癌、乳腺癌等多种癌症。
研究结果
|单细胞转录组测序揭示癌症细胞系内的潜在转录异质性
研究人员利用DNBelab C4获得了40个人类癌症细胞系和2个人类正常细胞系的23,089个细胞,平均每个细胞系513个细胞,包含34,641个转录本,每个细胞捕获了5,859个基因,显示了该数据集的高质量。之后通过Seurat将42个细胞系形成的不同簇进行UMAP可视化,绘制了单细胞转录组图谱(图1)。
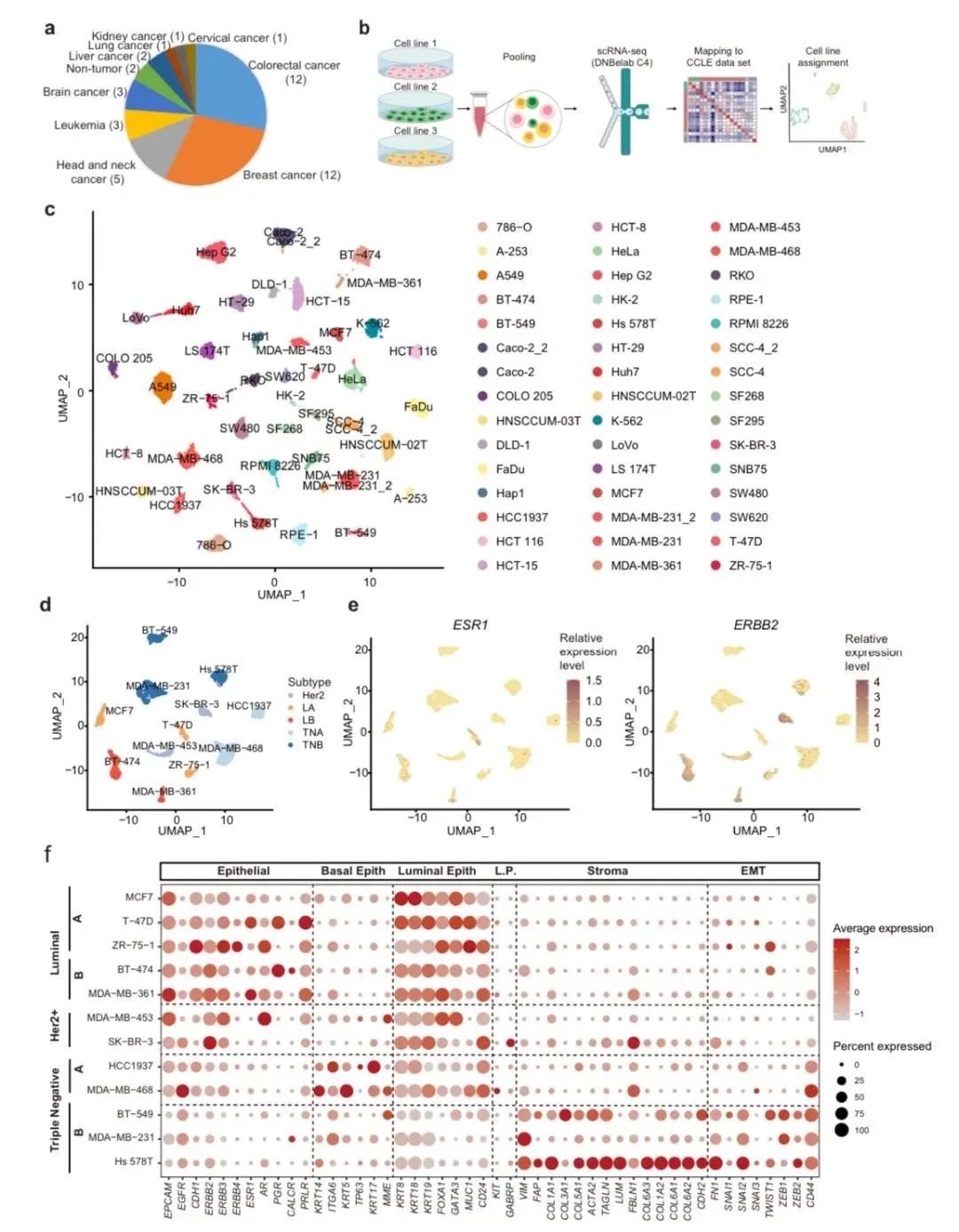
图1 scRNA-seq揭示细胞系内单细胞水平的异质性特征
研究人员进一步利用UMAP可视化的单细胞转录组图谱结合多样性评分,对癌症细胞系内异质性进行了量化,发现癌症细胞系内异质性是人类癌症细胞系的共同特征,但不同细胞系的异质性程度不同(图2)。此外,癌症细胞系内异质性可能受到不同亚克隆和细胞系内在可塑性的影响,不同细胞系之间的相对贡献有所差异。总之,这些发现揭示了即使在表面上看起来是同系的细胞,实际上存在着细胞系内的潜在异质性。
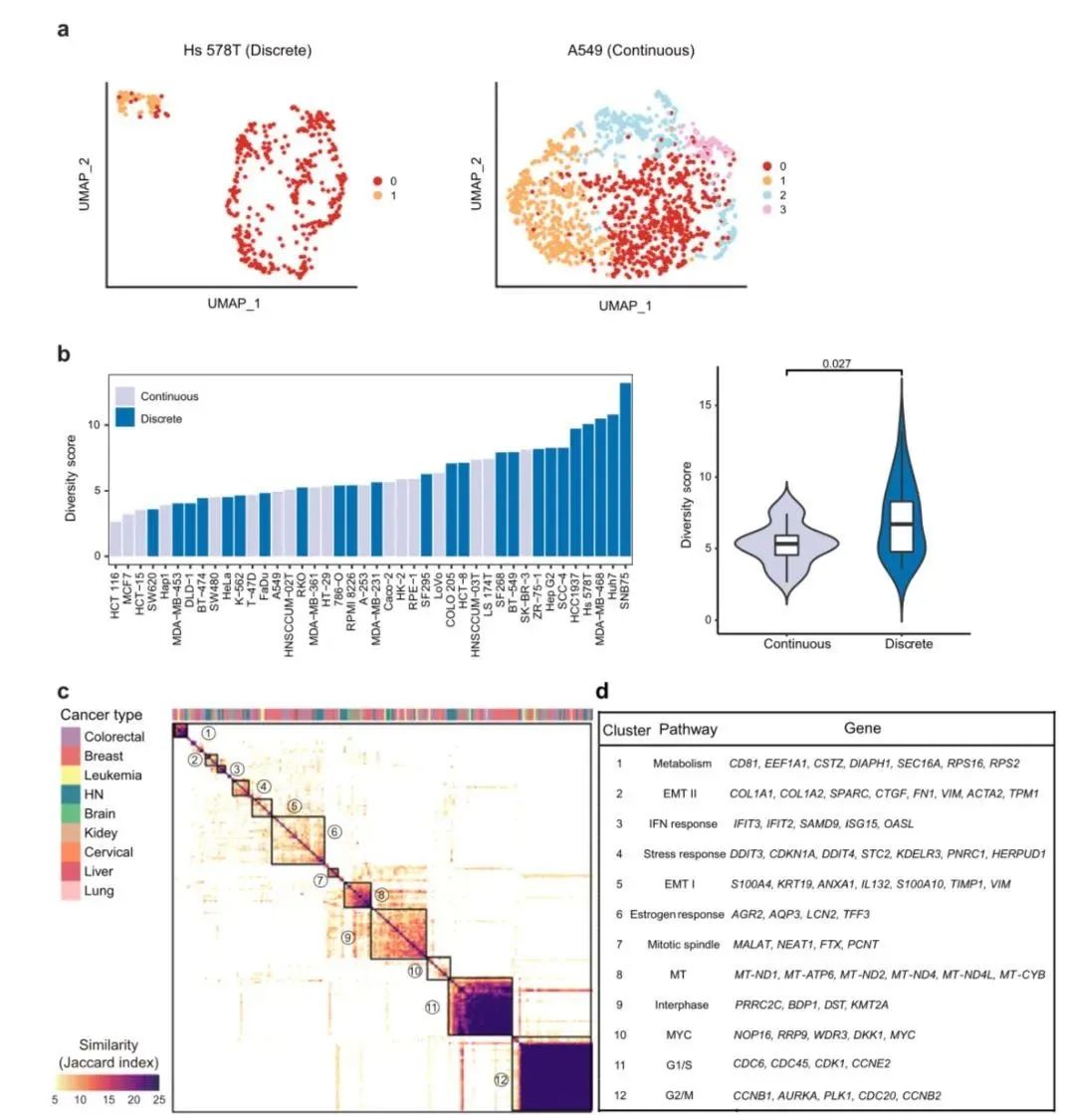
图2 细胞系内转录组异质性的不同模式
|单细胞转录组与表观基因组的相互作用
虽然单细胞转录组技术可以检测细胞的多样性,但仅仅基于转录组数据来解释表观基因组调控编码仍然是一项挑战。相比之下,scATAC-seq可以更直接地描述异质细胞群体中增强子和启动子的全基因组动态。因此,研究人员进一步利用scATAC-seq来研究单个细胞系的表观基因组异质性(图3)。
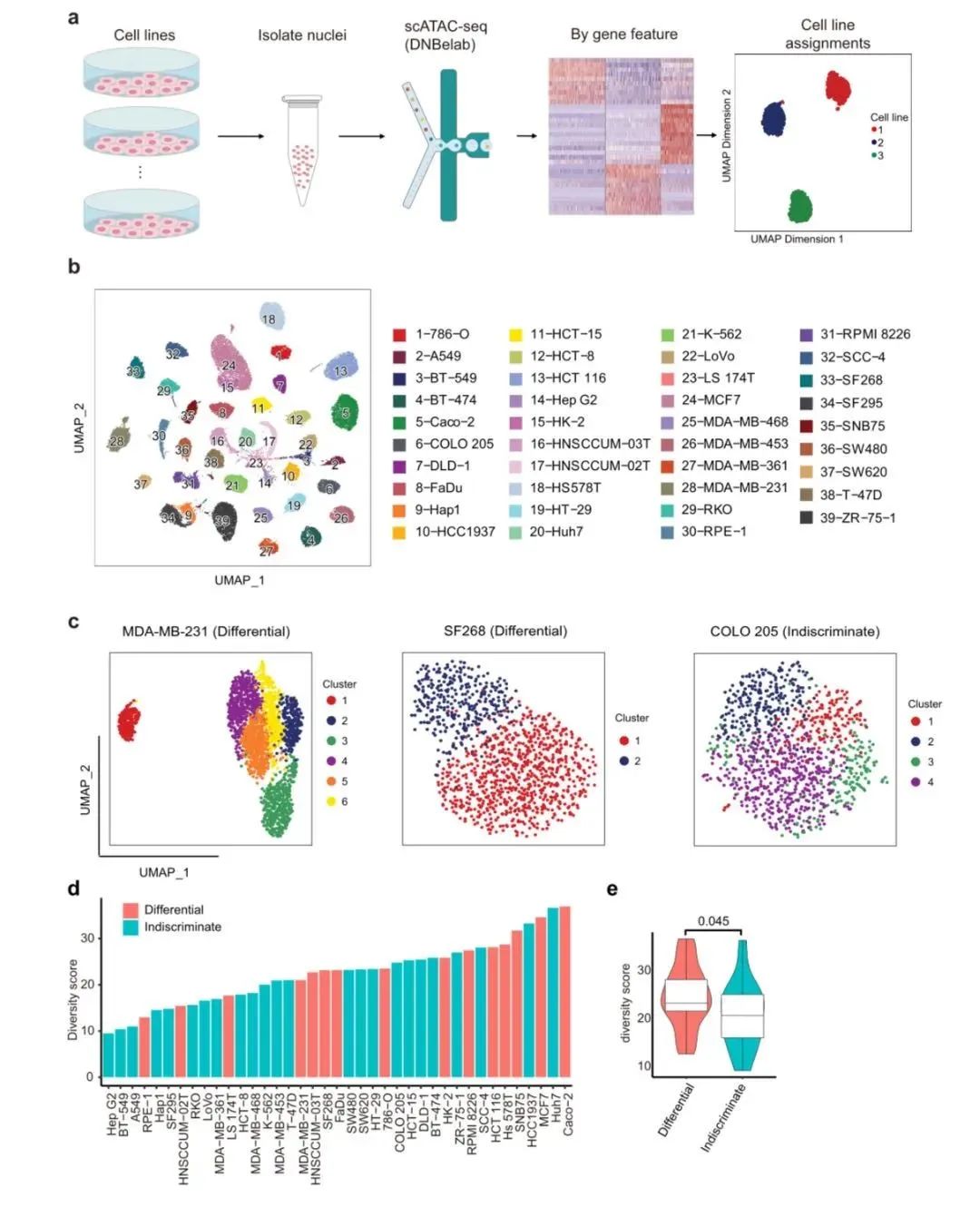
图3 scATAC-seq揭示细胞系内染色质可及性特征
为了确定观察到的转录异质性的来源,研究人员首先分析了恶性肿瘤中较为常见的基因变异。根据scRNA-seq数据推断了拷贝数变异(copy number variation,CNV),发现基因变异是决定离散模式细胞系内异质性的重要因素(图4)。然而,并不是所有的离散模式细胞系都有CNV亚克隆,而且CNV亚克隆并不总是与转录亚群相联系,表明有其他因素影响转录异质性。
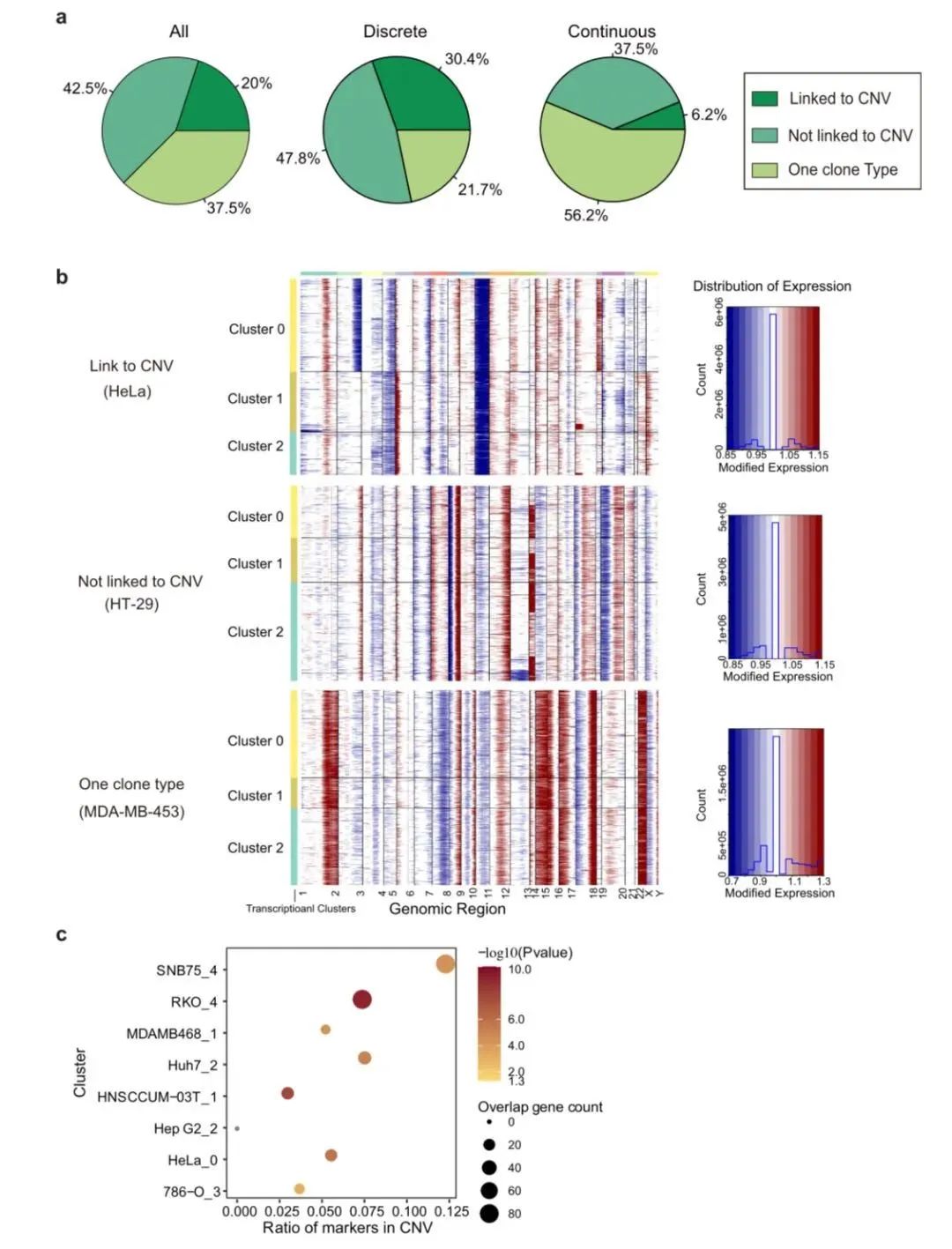
图4 CNVs在转录组异质性中的作用评估
为了进一步探索表观基因组异质性的机制,研究人员首先利用ArchR和ChromVAR工具确定了驱动亚簇之间染色质可及性差异的潜在转录因子(transcription factors,TFs),并指出FOXA2、NFE2L2和NFYB等TFs在驱动癌症细胞系异质性方面发挥着重要作用(图5)。
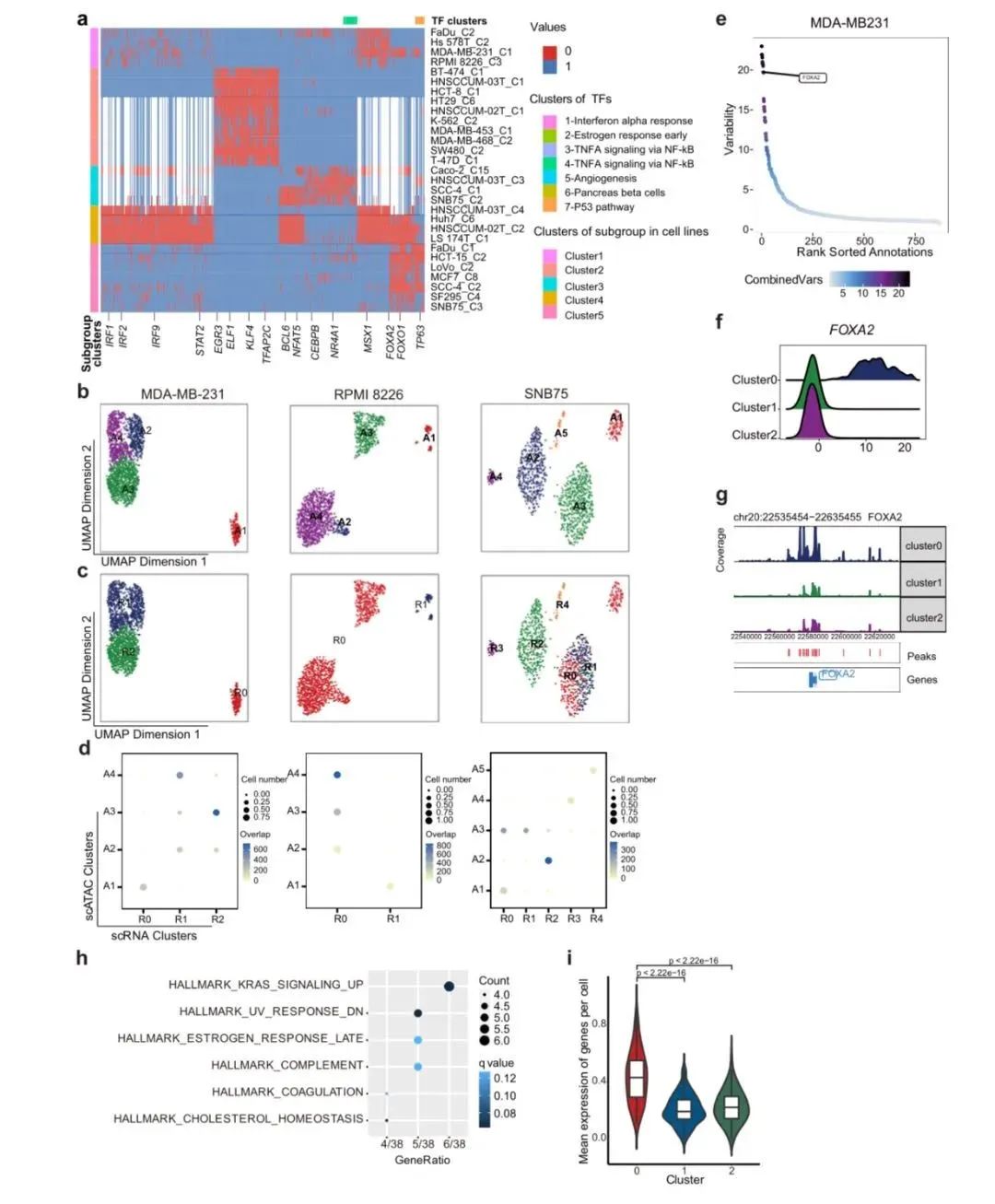
图5 受表观遗传可塑性调节的表达异质性
最近的研究表明,癌症基因可以在染色质外环状DNA(extrachromosomal circular DNAs,ecDNA)上扩增,因此研究人员进一步利用scATAC-seq数据检测了潜在的ecDNA区域,成功鉴定了经实验证实的ecDNA,并表明癌症基因在这些区域富集(图6)。研究结果表明,ecDNA可通过增加基因的拷贝数和影响特定TFs的活性来促进转录表达的异质性,并证明了ecDNA对多种细胞系(包括MDA-MB-231和SNB75)中细胞异质性的影响。
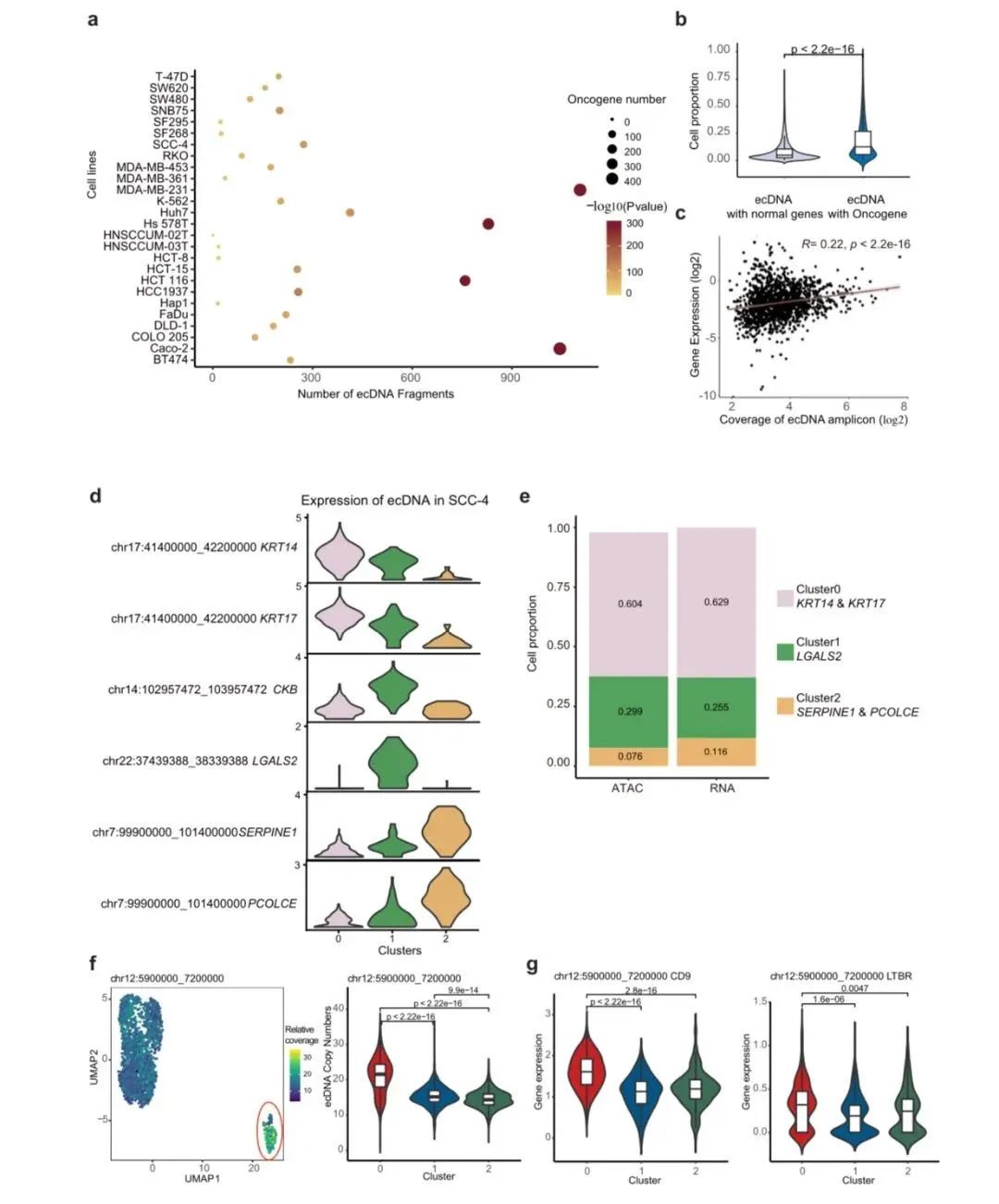
图6 ecDNA对细胞转录异质性的贡献
|细胞系增殖过程中的转录动态变化
研究人员进一步利用谱系追踪实验探讨了癌症细胞系中细胞异质性的动态变化。通过追踪MDA-MB-231和HCT 116细胞系中单细胞及其衍生细胞的独特核酸序列(分别代表离散和连续异质性模式),研究人员在不同时间点观察到稳定的细胞组成和相似的聚类模式,表明了总体分布的稳定性。然而,单个细胞具有显著的转录组可塑性,表现出在同一细胞群内的不同状态之间转换(图7)。这种现象在离散和连续细胞系中均被观察到,表明其在癌症细胞群中的广泛存在性。
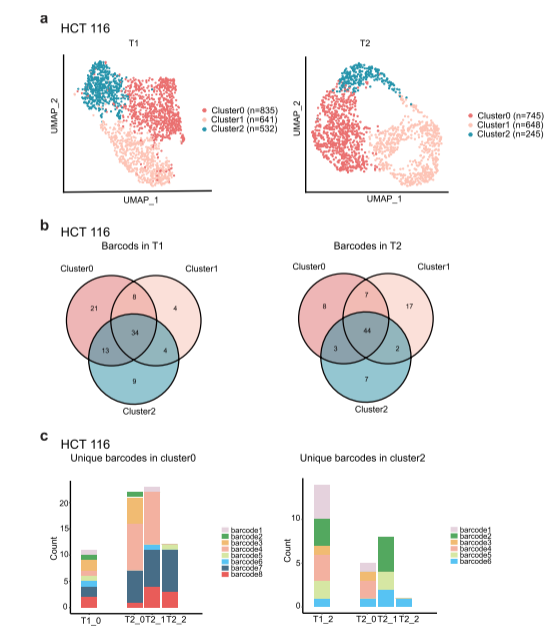
图7 单细胞转录组异质性的可塑性
|低氧环境驱动癌症细胞异质性响应
研究人员进一步评估了这些转录异质性在不同条件下是静态的还是可塑性的。由于实体瘤中肿瘤细胞往往处于不同程度的低氧状态,因此研究人员选择低氧干预作为环境应激条件,发现低氧会引起基因表达的显著变化,特别是与低氧相关的基因(图8)。研究人员观察到两种类型的细胞响应模式,一种是低氧前后细胞簇的一对一匹配,表明了稳定的异质性(以ZR-75-1为例);另一种是一些细胞簇从一个分裂到多个,表明同一群体内存在差异响应(以DLD-1、SW620为例)。这些发现表明,异质性染色质可及性促进了看似同系的细胞群的异质性应激响应。
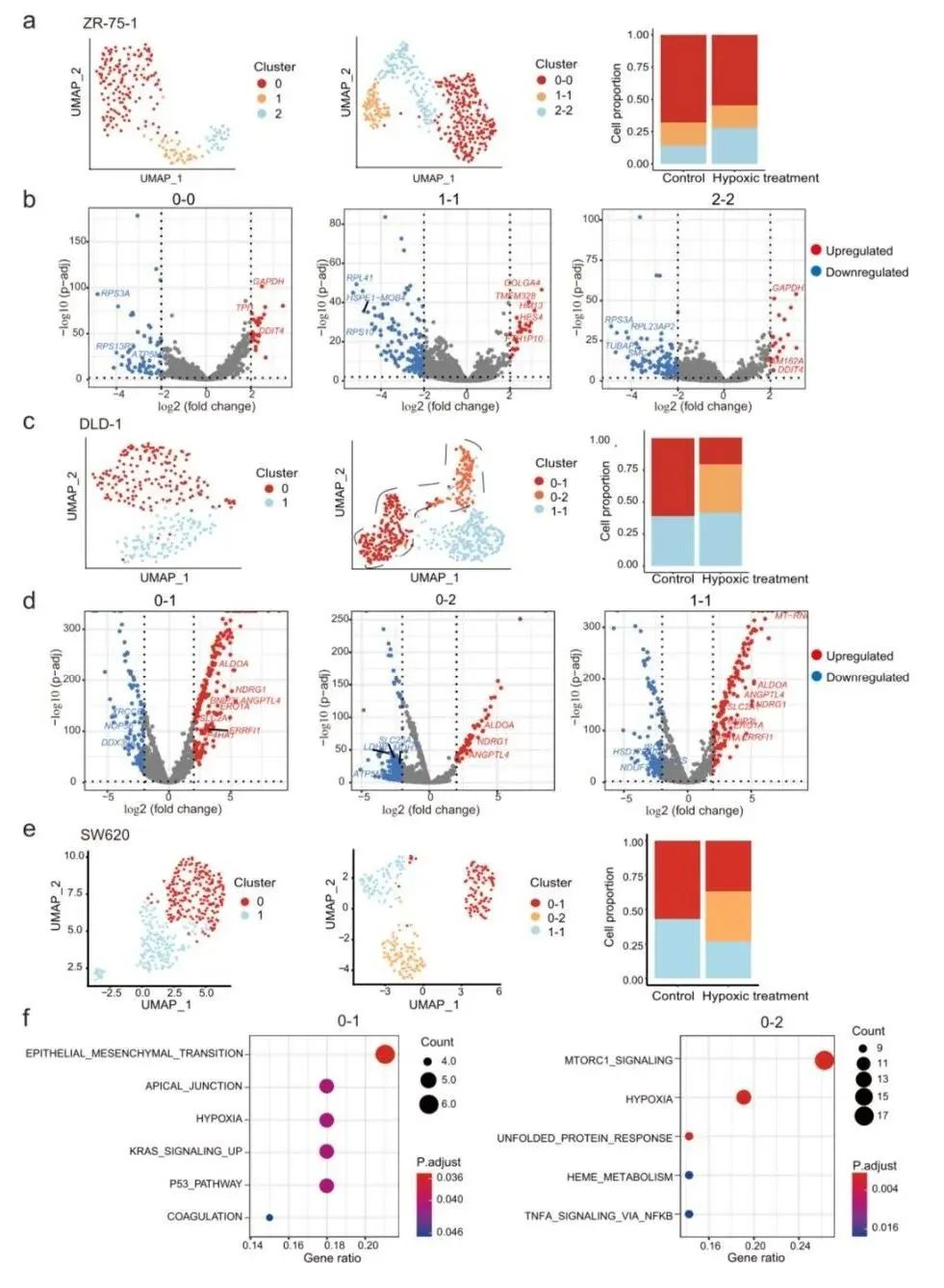
图8 细胞在低氧环境下的差异响应
研究结论
该研究通过单细胞转录组和表观基因组分析,成功揭示了在人类癌症细胞系中存在的异质性。研究发现,在表面上看起来是同系的细胞中,存在潜在的异质性,且在细胞系的扩增过程中,单个细胞的转录组特征会发生动态变化。通过整合单细胞转录组和单细胞表观基因组数据,研究显示转录组和表观基因组之间存在相互作用,此外,研究发现低氧干预可以驱动细胞的异质性响应。
这些发现为深入理解同一癌症细胞系的单细胞水平的异质性提供了重要的见解,并为制定个体化治疗策略和解决癌症治疗耐药性提供了新的思路和理论基础。
华大生命科学研究院吴靓研究员、刘石平研究员、刘龙奇研究员,南方科技大学陈炜教授、方亮副研究员为该论文共同通讯作者。南方科技大学朱琼花博士、李艳萍,华大生命科学研究院赵鑫、张远航、刘尚为论文共同第一作者。华大生命科学研究院院长徐讯、时空组学领域首席科学家陈奥等对该研究亦有贡献。
参考文献:
Zhu, Q., Zhao, X., Zhang, Y. et al. Single cell multi-omics reveal intra-cell-line heterogeneity across human cancer cell lines. Nat Commun 14, 8170 (2023). https://doi.org/10.1038/s41467-023-43991-9
信息及图片来源:“华大时空”公众号。
文章内容来源于“国家基因库大数据平台”公众号,本网站仅用于学术分享,如有侵权请联系管理员删除或修改。